EN ESTA UNIDAD NOS DIMOS CUENTA DE LA REPLICACION TANTO DE EUCARIOTAS COMO DE PROCARIOTAS, LA REPLICACION SE LLEBA ACABO DE FORMA SIMILAR EN PROCARIOTAS Y ECUCARIOTAS, LA REPLICACION ES MUY IMPORTANTE YA QUE POR MEDIO DE UNA MOLECULA DE ADN OBTENEMOS DOS EXACTAMENTE IGUALES CADA UNA CON UNA HEBRA NUEVA Y UNA HEBRA ANTIGUA POR ESTO ES QUE SE LE LLAMA A ESTE MODELO SEMICONSERVATIVO. ADEMAS DE QUE EN EL LABORATORIO LLEVAMOS ACABO UNA PRACTICA DE EXTRACCION DE ADN PARA NUETRO MEJOR ENTENDIMIENTO.
domingo, 18 de marzo de 2012
viernes, 16 de marzo de 2012
La reacción en cadena de la polimerasa, conocida como PCR por sus siglas en inglés (Polymerase Chain Reaction), es una técnica de biología molecular desarrollada en 1986 por Kary Mullis, cuyo objetivo es obtener un gran número de copias de un fragmento de ADN particular, partiendo de un mínimo; en teoría basta partir de una única copia de ese fragmento original, o molde.
Esta técnica sirve para amplificar un fragmento de ADN; su utilidad es que tras la amplificación resulta mucho más fácil identificar con una muy alta probabilidad, virus o bacterias causantes de una enfermedad, identificar personas (cadáveres) o hacer investigación científica sobre el ADN amplificado. Estos usos derivados de la amplificación han hecho que se convierta en una técnica muy extendida, con el consiguiente abaratamiento del equipo necesario para llevarla a cabo.
Fundamento e importancia
Esta técnica se fundamenta en la propiedad natural de los ADN polimerasas para replicar hebras de ADN, para lo cual emplea ciclos de altas y bajas temperaturas alternadas para separar las hebras de ADN recién formadas entre sí tras cada fase de replicación y, a continuación, dejar que vuelvan a unirse las polimerasas para que vuelvan a duplicarlas. La reacción en cadena de la polimerasa fue perfeccionada por Kary Mullis perteneciente a la Cetus Corporation en California SA, en la década de 1980.
Inicialmente la técnica era lenta, ya que las polimerasas se desnaturalizaban al realizar los cambios de temperatura y era necesario agregar nuevas polimerasas en cada ciclo. Puesto que las temperaturas del ciclo (95 °C en las fases de desnaturalización del ADN) suponen la inmediata desnaturalización de toda proteína, se emplean ADN polimerasas termoestables, extraídas de microorganismos adaptados a vivir a esas temperaturas, restrictivas para la mayoría de los seres vivos. Dichos microorganismos, generalmente arqueas, son: Thermus aquaticus (polimerasa Taq), Pyrococcus furiosus (Pfu), Thermococcus litoralis (Vent) y Thermus termophilus (Tth). Generalmente se emplean mezclas de polimerasas muy procesivas (Taq) con otras capaces de hacer corrección de errores (Pfu, Vent).
Hoy, todo el proceso de la PCR está automatizado mediante un aparato llamado termociclador, que permite calentar y enfriar los tubos de reacción para controlar la temperatura necesaria para cada etapa de la reacción (ver más abajo). Muchos termocicladores modernos hacen uso del efecto Peltier, que permite tanto calentar como enfriar los tubos simplemente invirtiendo la corriente eléctrica. Los tubos usados para PCR tienen una pared muy fina, lo que favorece una buena conductividad térmica, permitiendo que se alcance rápidamente el equilibrio térmico. Casi todos los termocicladores tienen un sistema que calienta la tapa de cierre con el fin de evitar la condensación sobre los tubos de reacción. Los termocicladores más antiguos que carecían de este sistema, solucionaban el problema de la condensación con una capa de aceite en la parte superior de la mezcla de reacción o con un poco de cera dentro de los tubos.
Por lo general, la PCR es una técnica común y normalmente indispensable en laboratorios de investigación médica y biológica para una gran variedad de aplicaciones. Entre ellas se incluyen la clonación de ADN para la secuenciación, la filogenia basada en ADN, el análisis funcional de genes, el diagnóstico de trastornos hereditarios, la identificación de huellas genéticas (usada en técnicas forenses y test de paternidad) y la detección y diagnóstico de enfermedades infecciosas.
Reactivos
- Los 4 desoxirribonucleósidos-trifosfato (dNTP), sustratos para polimerizar nuevo ADN.
- Dos cebadores o iniciadores (en inglés, primers), oligonucleótidos que son, cada uno, complementarios a una de las dos hebras del ADN. Son secuencias cortas, de entre seis y cuarenta nucleótidos, normalmente de 18 a 22, que permiten que la polimerasa inicie la reacción. Deben estar situados enfrentados y a no mucha distancia. Delimitan la zona de ADN a amplificar, es decir, corresponden a los nucleótidos que definen los extremos de la secuencia que se desea replicar.
- Iones divalentes. Se suele usar magnesio (Mg2+), agregado comúnmente como cloruro de magnesio (MgCl2), o algún otro catión divalente. También se puede emplear manganeso (Mn2+), para mutagénesis de ADN mediante PCR, ya que altas concentraciones de Mn2+ incrementan la tasa de error durante la síntesis de ADN. Actúan como cofactores de la polimerasa.
- Iones monovalentes, como el potasio.
- Una solución tampón (buffer) que mantiene el pH adecuado para el funcionamiento de el ADN polimerasa.
- ADN polimerasa o mezcla de distintas polimerasas con temperatura óptima alrededor de 70 °C (la más común es la polimerasa Taq).
- ADN molde, que contiene la región de ADN que se va a amplificar.
- Termociclador, el aparato que va a mantener la temperatura necesaria en cada una de las etapas que conforman un ciclo.
CONTROL GENETICO DE LA REPLICACION
El alcance deL conocimiento del ciclo celular ha sufrido una revolución en los últimos años. En el pasado, utilizando únicamente el microscopio óptico, los científicos dividieron el ciclo celular en dos periodos. Uno, de elevada actividad de reorganización y movimiento de los componentes subcelulares, denominado mitosis en el que los cromosomas se condensan haciendose visibles al microscopio optico y que finaliza con los procesos de segregacion y divicion celular. y un segundo que comprende la mayor parte del ciclo celular,denominado interfase por transcurrir entre dos mitosis y en el que a microscopio optico no se observa proceso activo alguno salvop el incremento en el tamaño celular.hoy se sabe que la interfase no es un periodo de reposo sino que por el contrario es un periodo de gran actividad, durante el que se llevan a cabo en una secuencia ordenada complicados y elaborados preparativos para la división celular.Efectivamente, durante la interfase y de forma continua tienen lugar acontecimientos conocidos en su globalidad como crecimiento y que incluyen la síntesis de nuevos orgánulos celulares y la mayor parte de las proteínas celulares. Además, comprende procesos (figura 2) que ocurren de forma discontinua, como la replicación del ADN durante el periodo conocido como
fase S -de Síntesis-. El intervalo que transcurre entre el final de la mitosis del ciclo anterior y el comienzo
de la síntesis de ADN se conoce como
fase G1 –del inglés Gap- y el intervalo entre
el fin de la fase S y la mitosis,
fase G2.
Las fases G1 y G2 proporcionan tiempo adicional para el crecimiento. Durante G1 la célula analiza si las condiciones ambientales son adecuadas para comenzar el proceso irreversible de la división celular, siempre que cuente con el tamaño mínimo para ello. En el caso de que la situación sea favorable, toma la decisión de atravesar el punto de inicio (start) que compromete irreversiblemente el comienzo del ciclo celular. si las condiciones ambientales no son adecuadas para la entradaen el ciclo, la célula entra en un periodo de quiescencia, denominado G0, en el que puede permanecer largo tiempo. Además, desde este estadío de G0 la célula es sensible a señales de diferenciación, en el caso de organismos pluricelulares, o de inicio de procesos sexuales, en el caso de organismos unicelulares. Si las condiciones ambientales vuelven a ser favorables, la célula puede volver a iniciar ciclos de división. En la fase G2 la célula analiza si ha completado correctamente la fase S y decide entre permitir el paso a mitosis, o en su caso, esperar para que se realicen las reparaciones necesarias.
CONTROL DEL CICLO CELULAR
La mayor parte de los estudios y los primeros avances sobre los mecanismos de control del ciclo celular en eucariotas se han realizado empleando como modelo las levaduras. Las levaduras son hongos unicelulares que presentan grandes ventajas como modelo de estudio del ciclo celular eucariota. Son organismos muy adecuados porque tienen tiempos de generación cortos, (90 minutos frente a las 24 horas que dura el ciclo de las células animales) y su genoma es alrededor de 100 veces menos complejo que el de una célula de mamífero, pero mantienen el mismo tipo de organización del ciclo celular que los eucariotas multicelulares. Además, es fácil de manipular genéticamente, siendo posible aislar mutaciones en genes que controlan procesos celulares básicos, clonar los genes identificados por esas mutaciones y realizar deleciones de genes específicos, al ser las levaduras capaces de vivir como organismos haploides.
La mayor parte de los estudios de ciclo celular se han llevado a cabo empleando
S. Serevisiae la levadura del pan y la cerveza también conocida como la levadura de gemación por su característico estilo de división. En el caso de este organismo hay que añadir la ventaja de que actualmente se conocen los aproximadamente 16 millones de pares de nucleótidos que constituyen la secuencia completa de su genoma.
Ciencia al Día © Febrero 2000, Vol. 3, No. 1. ISSN 0717-3849
REPLICACION EN EUCARIOTES
Es similar a la de los procariontes, es decir, semiconservativa y bidireccional. Existe una hebra conductora y una hebra retrasada con fragmentos de Okazaki. Se inicia en ORI (puede haber unas 100 a la vez), entre las diferencia, se comienza con las polimerasas, son más complejas, y además, la polimerasa de la hebra continua es diferente a la de la hebra discontinua. De la hebra continua se encarga la polimerasa Delta y de la discontinua, la Alfa.
Las helicasa difieren en estructura, las primasas se encuentran adosadas a la ADN-pol Alfa.
El resto del proceso es muy parecido.
En el final de la hebra antigua que da la base a la hebra discontinua, queda una zona denominada telómero, que no tiene como obtener su dupla en la hebra discontinua, debido a que no es posible insertar un nuevo ARN cebador, ni mucho menos un fragmento de Okazaki. Al no tener su par, son eliminados automáticamente, perdiendose. Existen enzimas, las telomerasas, que repiten esta secuencia varias veces, cosa que no se pierda información esencial del genoma. Las enzimas encargadas de cortar el telómero, son las topoisomerasas 4.
El ARN cebador es retirado por el complejo de reparación de la célula.
lunes, 12 de marzo de 2012
REPLICACION EN PROCARIOTES
Ocurre en tres etapas:
1ª etapa: desenrrollamiento y apertura de la doble hélice en el punto ori.
1ª etapa: desenrrollamiento y apertura de la doble hélice en el punto ori.
En el punto de origen u ORI, que es un lugar del cromosoma con gran contenido de A y T, la doble hélice se abre, mediante la DNA helicasa y las proteínas desestabilizadoras de la hélice o proteínas de unión a DNA de una sola cadena, vuelven recta la cromatina y la mantienen abierta. La DNA polimerasa sintetiza las cadenas complementarias a cada una de las cadenas primitivas. Forma dos copias activas de ADN, una es continua, o sea, basta con agregar los nucleotidos correspondientes porque la hebra antigua tiene 3', por lo que se crea una 5´. En la otra hebra, se produce un proceso discontinuo, debido a que la hebra quedo con un final 5', debiendo partir con un 3', y la célula es incapaz de seguir la cadena con este final, para que se inicie la copia del DNA hace falta un corto RNA específico (10 pares de bases), denominado RNA cebador, que hace que empiece a actuar la DNA polimerasa. El RNA cebador es generado por la RNA primasa (sintetizadora de RNA). Esta enzima se une directamente a la DNA helicasa, formando un complejo llamada primosoma, que se va desplazando con la cadena en formación. Conforme van existiendo fragmentos de cadena abiertos de suficiente longitud, se va sintetizando la cadena discontinua formando pequeños fragmentos, denominados Fragmentos de Okazaki, cada uno de unos 1000 nucleótidos. . Hace falta un RNA cebador por cada fragmento de Okazaki. La RNA primasa, va sintezando a intervalos los RNA cebadores que van siendo incorporados a la copia como si fueran ADN, entre los fragmentos de Okazaki, hasta que se alcanza el RNA cebador del fragmento de Okazaki ya terminado. . La cadena con ARN cebador, es denominada cadena retrasada
El DNA sintetizado en la cadena retrasada y su cadena patrón sufren un plegamiento, de tal forma que la DNA polimerasa de la cadena conductora se unen para formar un complejo único, de modo que las proteínas de replicación puedan utilizarse conjuntamente en la replicación de ambas cadenas. Las topoisomerasas mantienen esta estructura y evitan que el DNA se enrede por superenrrollamiento, cortando un enlace fosfodiester, y a esto se le llama nick.Además, existe una proteína llamada SSB, que estabiliza la forma monocatenaria de la hebra en replicación, para que no se acople por complementariedad de bases con ella misma.
Las enzimas ligasas son las encargadas, despues, de ir arreglando los nicks cuando se sustituye el ARN cebador.
Exiten tres tipos de ADN-polimerasa, la I, es la encarga de reparar la hebra; la II, de ayudar a la III; y la III, agrega las bases a la hebra.
La labor de la ADN-pol I es exonucleasa, puede poner bases como sacar bases, con esto puede reparar la hebra. El dominio de esta proteína encargado de incluir bases a la hebra se llama Fragmento Klenow, que puede ser liberado del resto de la proteína por la Tripsina.
jueves, 8 de marzo de 2012
REPLICACION DEL ADN
La replicación del ADN produce una copia de sí mismo por medio de enzimas que además de ser muy exactas poseen un sistema de reparación de errores. El mecanismo de replicación es es esencialmente el mismo en todas las células. Es un proceso semiconservativo porque cada uno de los dos ADN hijos tiene una cadena del ADN anterior.
martes, 6 de marzo de 2012
TAREA (EXPERIMENTO DE MESELSON Y STAHL
En cada una de las moléculas hijas se conserva una de las cadenas originales, y por eso se dice que la replicación del ADN es semiconservadora. Hasta que finalmente se pudo demostrar que la replicación es semiconservadora, se consideraron tres posibles modelos para el mecanismo de la replicación:
- Semiconservadora (modelo correcto). En cada una de las moléculas hijas se conserva una de las cadenas originales.
- Conservadora. Se sintetiza una molécula totalmente nueva, copia de la original.
- Dispersora, o dispersante. Las cadenas hijas constan de fragmentos de la cadena antigua y fragmentos de la nueva.
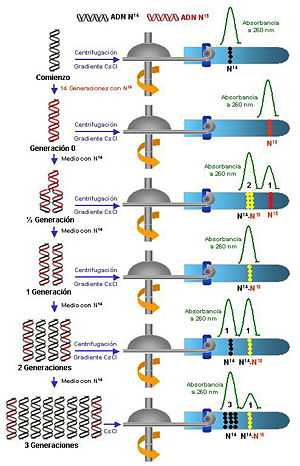
El experimento de Meselson y Stahl en 1958 permitió demostrar que el mecanismo real se ajusta a la hipótesis de replicación semiconservadora. Para ello se hicieron crecer células de Escherichia coli en presencia de nitrógeno-15, un isótopo del nitrógeno más pesado de lo habitual. En consecuencia, el isótopo se incorporó a las cadenas de ADN que se iban sintetizando, haciéndolas más pesadas.
Una vez conseguido el primer objetivo, las células fueron transferidas a un medio que contenía nitrógeno-14, es decir, un medio más ligero, donde continuaron su crecimiento (división celular, que requiere la replicación del ADN). Se purificó el ADN y se analizó mediante una centrifugación en gradiente de cloruro de cesio, en donde hay más densidad en el fondo del tubo que en la parte media del mismo.
En la primera generación se obtuvo una única banda de ADN con densidad intermedia. En la segunda generación se obtuvieron dos bandas, una con densidad ligera y otra con densidad intermedia o híbrida. En la tercera generación se obtuvieron dos bandas, una ligera (con una abundancia del 75%) y otra intermedia (con el 25% restante).
La banda intermedia o híbrida representa una molécula de ADN que contiene una cadena pesada (original) y otra ligera (recién sintetizada). Las cadenas ligeras representan una molécula de ADN en la que las dos cadenas han sido sintetizadas (no existían aun cuando las células se pusieron en presencia de nitrógeno-15.
El hecho de que cada vez haya más moléculas ligeras y se mantenga el número de moléculas intermedias demuestra que la replicación del ADN es semiconservadora. Si fuera conservadora, aparecería siempre una banda pesada y el resto ligeras. Si fuera dispersante sólo aparecerían bandas híbridas de densidad intermedia en todas las generaciones.
INTRODUCCION
REPLICACION DEL ADN
Se produce siempre en sentido 5' → 3', siendo el extremo 3'-OH libre el punto a partir del cual se produce la elongación del ADN. Esto plantea un problema, y es que las cadenas tienen que crecer simultáneamente a pesar de que son antiparalelas, es decir, que cada cadena tiene el extremo 5' enfrentado con el extremo 3' de la otra cadena. Por ello, una de las cadenas debería ser sintetizada en dirección 3' → 5'.
Este problema lo resolvieron los científicos japoneses Reiji Okazaki y Tsuneko Okazaki en la década de 1960, al descubrir que una de las nuevas cadenas de ADN se sintetiza en forma de trozos cortos que, en su honor, se denominan fragmentos de Okazaki. Su longitud suele variar entre 1000 y 2000 nucleótidos en las bacterias y entre 100 y 400 nucleótidos en eucariontes.
 La cadena que se sintetiza en el mismo sentido que avanza la horquilla de replicación se denomina hebra adelantada (en inglés, leading strand, que a veces se traduce por líder o conductora) y se sintetiza de forma continua por la ADN polimerasa, mientras que la que se sintetiza en sentido contrario al avance se denomina hebra rezagada o retrasada (en inglés, lagging strand), cuya síntesis se realiza de forma discontinua teniendo que esperar a que la horquilla de replicación avance para disponer de una cierta longitud de ADN molde.
La cadena que se sintetiza en el mismo sentido que avanza la horquilla de replicación se denomina hebra adelantada (en inglés, leading strand, que a veces se traduce por líder o conductora) y se sintetiza de forma continua por la ADN polimerasa, mientras que la que se sintetiza en sentido contrario al avance se denomina hebra rezagada o retrasada (en inglés, lagging strand), cuya síntesis se realiza de forma discontinua teniendo que esperar a que la horquilla de replicación avance para disponer de una cierta longitud de ADN molde.La helicasa rompe los puentes de hidrógeno de la doble hélice permitiendo el avance de la horquilla de replicación.
La topoisomerasa impide que el ADN se enrede debido al superenrollamiento producido por la separación de la doble hélice.
Las proteínas SSB se unen la hebra discontínua de ADN, impidiendo que ésta se una consigo misma.
La ADN polimerasa III sintetiza la cadena complementaria de forma continua en la hebra adelantada y de forma discontínua en la hebra rezagada.
La ARN primasa sintetiza el cebador de ARN necesario para la síntesis de la cadena complementaria a la cadena rezagada.
La ADN ligasa une los fragmentos de Okazaki.
El cebador: son pequeñas unidades de RNA que se unen a los fragmentos para que la ADN polimerasa reconozca donde debe unirse.
La ADN ligasa une los fragmentos de Okazaki.
El cebador: son pequeñas unidades de RNA que se unen a los fragmentos para que la ADN polimerasa reconozca donde debe unirse.
INICIACIÓN
ELONGACIÓN
los cebadores los quita la ADN polimerasa I y coloca bases a la cadena en crecimiento por la ligasa mediante consumo de ATP en dirección a la horquilla de replicación, es decir, en dirección 5' → 3' en la hebra rezagada y 3' → 5' en la hebra adelantada, rompiendo los puentes de hidrógeno que mantienen unida la doble hélice.[3] El siguiente conjunto de proteínas reclutadas son las denominadas proteínas SSB (single-stranded DNA binding proteins, proteínas ligantes de DNA monocatenario) encargadas de la estabilización del ADN monocatenario generado por la acción de las helicasas, impidiendo así que el ADN se renaturalice o forme de nuevo la doble hélice, de manera que pueda servir de molde. Estas proteínas se unen de forma cooperativa, por lo que su unión al DNA conforme avanza la helicasa es rápida. Por otro lado, conforme las helicasas van avanzando se van generando superenrollamientos en la doble cadena de ADN por delante de la horquilla y si éstos no fueran eliminados, llegado a un punto el replisoma ya no podría seguir avanzando. Las topoisomerasas son las enzimas encargadas de eliminar los superenrollamientos cortando una o las dos cadenas de ADN y pasándolas a través de la rotura realizada, sellando a continuación la brecha
ELONGACIÓN
Enzimas que participan en la replicación de E. coli: helicasa, proteínas SSB, topoisomerasa, ARN primasa, Holoenzima ADN Pol IIIEn el siguiente paso, la holoenzima ADN Pol III cataliza la síntesis de las nuevas cadenas añadiendo nucleótidos sobre el molde. Esta síntesis se da bidireccionalmente desde cada origen, con dos horquillas de replicación que avanzan en sentido opuesto. Cuando el avance de dos horquillas adyacentes las lleva a encontrarse, es decir, cuando dos burbujas se tocan, se fusionan, y cuando todas se han fusionado todo el cromosoma ha quedado replicado.
Puesto que la holoenzima ADN Pol III necesita de un extremo 3'-OH libre, es necesario que una ARN primasa catalice la formación de un fragmento corto específico de ARN llamado cebador, que determinará el punto por donde la ADN polimerasa comienza a añadir nucleótidos. Así, durante la síntesis, en cada horquilla de replicación se van formando dos copias nuevas a partir del cebador sintetizado en cada una de las dos hebras de ADN que se separaron en la fase de iniciación, pero debido a la unidireccionalidad de la actividad polimerasa de la ADN Pol III, que sólo es capaz de sintetizar en sentido 5´ → 3', la replicación sólo puede ser continua en la hebra adelantada; en la hebra rezagada es discontinua, dando lugar a los fragmentos de Okazaki.
La mitad del dímero de la holoenzima ADN Pol III sintetiza la hebra adelantada y la otra mitad la hebra rezagada.
En la hebra rezagada, cuando la ADN Pol III hace contacto con el extremo de otro fragmento de Okazaki contiguo, el cebador de ARN de éste es eliminado y los dos fragmentos de Okazaki de ADN recién sintetizado son unidos. Una vez se han juntado todos se completa la doble hélice de ADN. La eliminación de cebadores también se da en la hebra conductora, de síntesis continua, pero debido a que en ésta hay un solo cebador es un proceso que sólo tiene lugar una vez, mientras que en la hebra rezagada se dará tantas veces como fragmentos de Okazaki haya. Para ello intervienen una serie de enzimas: la enzima RNasa H elimina el cebador a excepción del ribonucleótido directamente unido al ADN; la ADN Pol I elimina este ribonucleótido gracias a su actividad exonucleasa 5' → 3' y rellena el hueco con ADN quedando una molécula completa a excepción de una rotura (o "mella") entre el extremo 3'-OH libre y el fosfato 5' de la cadena reparada; por último, la ADN ligasa sella esa rotura catalizando la reacción de condensación entre el grupo fosfato y el OH de la desoxirribosa del nucleótido contiguo, completando el enlace fosfodiéster; para ello, es preciso hidrolizar una molécula de ATP.
OBJETIVOS
Entender como se transmite la informacion genetica entre los seres vivos y su relacion con los mecanismos de herencia.
Distinguir los mecanismos de duplicacion del material genetico en procariotas y eucariotas.
domingo, 4 de marzo de 2012
CONCLUCIONES
en esta unidad nos dimos cuenta de como se encuentra organizado el mateial genetico tanto de organismos eucarioticos como de procarioticos, asi como las diferencias que se encuentran entre estos dos, por ejemplo : PROCARIOTICOS: poseen 1 cromosoma, el ADN es circular, es haploide.
EUCARIOTICOS : poseen varios cromosomas, el ADN el lineal, es diplode.
ademas de conocer tambien de como se lleba acabo el empaquetamiento del material genetico,asi como tambien conocimos las partes por las que se encuentra constituido un gen.
EUCARIOTICOS : poseen varios cromosomas, el ADN el lineal, es diplode.
ademas de conocer tambien de como se lleba acabo el empaquetamiento del material genetico,asi como tambien conocimos las partes por las que se encuentra constituido un gen.
jueves, 1 de marzo de 2012
ORGANIZACION GENOMICA VIRAL
Se puede ver una enorme variedad de estructuras genómicas entre las especies de virus que, como grupo, contienen una diversidad genómica superior a la de los reinos de plantas, los animales o las bacterias. Hay millones de diferentes tipos de virus; y únicamente alrededor de 5.000 de ellos han sido descritos detalladamente.
Un virus tiene un genoma compuesto de ADN o bien de ARN, y reciben respectivamente los nombres de «virus ADN» y «virus ARN». La gran mayoría de virus utilizan el ARN. Los virus de las plantas tienden a tener ARN monocatenario y los bacteriófagos tienden a tener ADN bicatenario.
Los genomas víricos pueden ser circulares, como los polyomaviridae o lineales, como los adenoviridae. El tipo de ácido nucleico es irrelevante para la forma del genoma. En los virus ARN, el genoma a menudo está dividido en partes separadas dentro del virión, y se le califica de segmentado. Cada segmento suele codificar una proteína y los segmentos suelen estar reunidos en una cápside. No es necesario que cada segmento se encuentre en el mismo virión porque el virus en general es infeccioso, como lo demuestra el Brome mosaic virus.
En los virus ARN o los virus ADN monocatenarios, las cadenas pueden ser o bien positivas (cadenas plus) o negativas (cadenas minus), dependiendo de si son complementarias en el ARN mensajero (ARNm) vírico. El ARN viral positivo es idéntico al ARNm viral y por tanto puede ser traducido inmediatamente por la célula huésped. El ARN viral negativo es complementario del ARNm y por tanto debe ser convertido en ARN positivo por una ARN polimerasa antes de ser traducido. La nomenclatura del ADN es similar a la del ARN, en cuanto a la «cadena codificadora» del ARNm vírico que le es complementaria (-), y la «cadena no codificadora» que es una copia (+).
El tamaño del genoma varía mucho entre especies. Los genomas víricos más pequeños sólo codifican cuatro proteínas y pesan unos 106 daltons; los más grandes pesan unos 108 daltons y codifican más de un centenar de proteínas. Los virus ARN suelen tener genomas más pequeños que los virus ADN debido a una tasa de error más alta a la hora de replicarse, y tienen un límite superior de tamaño. Por encima de este límite, los errores en la replicación del genoma hacen que el virus sea inofensivo o incluso, incompetente. Para compensar esto, los virus ARN a menudo inician un proceso de segmentación en el que el genoma es separado en moléculas más pequeñas, reduciendo así las posibilidades de error. En cambio, los virus ADN tienen genomas mayores gracias a la elevada fidelidad de sus enzimas de replicación.
| Propiedad | Parámetros |
|---|---|
| Ácido nucleico |
|
| Forma |
|
| Cadenas |
|
| Sentido |
|
El ADN mitocondrial es el material genético de las mitocondrias, los orgánulos que generan energía para la célula. El ADN mitocondrial se reproduce por sí mismo semi-autónomamente cuando la célula eucariota se divide. El ADN mitocondrial fue descubierto por Margit M. K. Nass y Sylvan Nass utilizando microscopia electrónica y un marcador sensitivo al ADN mitocondrial. Evolutivamente el ADN mitocondrial desciende de genomas circulares pertenecientes a bacterias, que fueron englobadas por un antiguo ancestro de las células eucarióticas.
Este ADN, al igual que los ADN bacterianos, es una molécula bicatenaria, circular, cerrada, sin extremos (cromosoma mitocondrial). En los seres humanos tiene un tamaño de 16.569 pares de bases, conteniendo un pequeño número de genes, distribuidos entre la cadena H (de heavy, pesada en inglés y la cadena L (de light, ligera), debido a su diferente densidad cuando son centrifugadas en gradiente de CsCl.
El número de genes en el ADN mitocondrial es de 37, frente a los 20.000 - 25.000 genes del ADN cromosómico nuclear humanos. Codifica dos ARN ribosómicos, 22 ARN de transferencia y 13 proteínas que participan en la fosforilación oxidativa. El cromosoma mitocondrial se organiza en "nucleoides", de tamaño variable y de unos 0,068 nanómetros de tamaño en humanos, y formados por entre 5-7 cromosomas y algunas proteínas, como el factor de transcripción mitocondrial A, la proteína de unión a ADN mitocondrial de cadena sencilla y la helicasa Twinkle. Su número por mitocondria es muy variable, pero su distribución se realiza a intervalos fijos, y muchos de ellos parecen localizarse en los "tubos mitocondriales". Parece ser que los nucleoides mitocondriales podrían tener un comportamiento "en capas", llevando a cabo la replicación en su centro, mientras que en la periferia sitúan la traducción de las proteínas necesarias para la cadena respiratoria. El número de tales nucleoides sería de varios cientos (400-800) en células de cultivo, y mucho menores en otras especies en que su tamaño es mayor.
El ADN mitocondrial está en replicación constante, independientementemente del ciclo y del tipo celular. Se piensa que tiene lugar de forma asíncrona, es decir, que tiene lugar en las dos cadenas en tiempos diferentes y con dos orígenes distintos hacia direcciones contrarias. El comienzo tendría lugar en el origen de la cadena pesada, situado en el bucle D, y replicaría ésta tomando como molde la cadena ligera. Cuando se alcanza el segundo origen, situado a dos tercios de distancia del primero, comienza la segunda ronda de replicación en sentido opuesto. Se ha propuesto un nuevo sistema de replicación que coexistiría con el primero. Sería bidireccional y comportaría una coordinación entre hebras directas y retrasadas. En la replicación en mamíferos estarían involucradas la polimerasa γ y la helicasa twinkle.
El ADN mitocondrial está sometido a un importante estrés por su proximidad con los centros de producción de radicales libres de oxígeno, de forma que disponen de una varia y compleja maquinaria de reparación, lo cual incluye diversas formas de recombinación, tanto homóloga como inhomóloga.
El genoma mitocondrial de los eucariotas se originó probablemente tras la endocitosis de una eubacteria aeróbica y la subsecuente transferencia sucesiva de muchos genes hacia el genoma nuclear.
Esta hipótesis surgió debido a que la organización del genoma mitocondrial es radicalmente diferente del genoma nuclear. Los genomas mitocondriales presentan varias características de los genomas procariotas como:
- Pequeño tamaño.
- Ausencia de intrones.
- Porcentaje muy elevado de ADN codificante.
- Falta generalizada de secuencias repetidas y genes de rARN comparativamente pequeños, parecidos a los de procariotas.
La evolución del código genético mitocondrial es probablemente el resultado de una presión de selección reducida en respuesta a una capacidad codificante muy disminuida.
Representación del ADN mitocondrial mostrando los loci afectados en algunas enfermedades humanas.
COMPLEJIDAD DEL GENOMA
El genoma es la totalidad de la información genética que posee un organismo en particular y que codifica para él. Por lo general, al hablar de genoma en los seres eucarióticos nos referimos sólo al ADN contenido en el núcleo, organizado en cromosomas. Pero no debemos olvidar que también la mitocondria contiene genes.
El término fue acuñado en 1920 por Hans Winkler, profesor de Botánica en la Universidad de Hamburgo, Alemania, como un acrónimo de las palabras gene y chromosoma.
El término diploide indica que un organismo tiene dos copias del genoma en sus células, debido a la presencia de pares de cromosomas homólogos.
El genoma no analiza la diversidad genética o el polimorfismo de los genes de una especie. Por ejemplo, en el genoma humano la secuencia en principio podría ser determinada con sólo la mitad del ADN de una célula de un individuo. Para conocer una variación particular o en enfermedades se requiere la comparación entre individuos mediante el genotipado.
CROMOSOMAS
Los cromosomas son el mayor grado de empaquetamiento que alcanza las moléculas de ADN.
Cada especie tiene un número determinado de cromosomas y de cierta froma propia de su especie. Así por ejemplo todos los perros poseen 78 cromsomas y además son todos de la misma morfología.
Los cromosomas tienen diversas partes que se muestran en el esquema.
Cada especie tiene un número determinado de cromosomas y de cierta froma propia de su especie. Así por ejemplo todos los perros poseen 78 cromsomas y además son todos de la misma morfología.
Los cromosomas tienen diversas partes que se muestran en el esquema.
Las distintas morfologías se definen segun el centrómero los divide en dos brazos. Cuando ambos brazos son iguales de denominan METACENTRICOS. Cuando un brazo es un poco más pequeño (brazo p) se denominan SUBMETACENTRICOS. El brazo más largo se llama q. En el caso que el brazo p sea muy pequeño se denominan SUBTELOCENTRICOS. En cambio cuando es casi imperceptible al microscopio sellaman ACROCÉNTRICOS. Si no hay brazo p y solo exite el brazo q se denominan TELOCÉNTRICOS.
En cambio en los cerdos existen 38 cromosomas de diferentes morfologías.
Todas las especies superiores son diploides es decir que heredan un juego de cromosomas de su madre y otro igual del padre. Por lo tanto hay dos cromosomas homólogos de cada uno de los totales.
El análisis de los cromosomas de un individuo se realiza mediante una técnica denominada CARIOTIPO que se base en obtener una foto de cromosomas en Metafase de la división celular,en dónde alcanza mayor estado de compactamiento. Generalmente luego se realiza el bandeo cromosoómico para ayudar a identificar a los cromosomas ya que cada uno tiene un patron de bandas propio de ese cromosoma en cada una de las especies difrentes.
Luego de obtenida la foto, se amplía y recorta cada uno de los cromosomas y se los ordena de mayor a menor tamaño dentro de cada morfología.
El análisis de los cromosomas de un individuo se realiza mediante una técnica denominada CARIOTIPO que se base en obtener una foto de cromosomas en Metafase de la división celular,en dónde alcanza mayor estado de compactamiento. Generalmente luego se realiza el bandeo cromosoómico para ayudar a identificar a los cromosomas ya que cada uno tiene un patron de bandas propio de ese cromosoma en cada una de las especies difrentes.
Luego de obtenida la foto, se amplía y recorta cada uno de los cromosomas y se los ordena de mayor a menor tamaño dentro de cada morfología.
Exiten nuevas técnicas para visualizar mejor a los cromosomas como el FISH o Chromosome Painting.
SOLENOIDE
El primer enrrollamiento que sufre el ADN por las histonas se denomina solenoide.
La cromatina en el núcleo tiene un grosor de 300Å. La fibra de cromatina de 100Å se empaqueta formando una fibra de cromatina de 300Å. El enrollamiento que sufre el conjunto de nucleosomas recibe el nombre de solenoide.
Los solenoides se enrollan formando la cromatina del núcleo interfásico de la célula eucariota. Cuando la célula entra en división, el ADN se compacta más, formando los cromosomas.
HISTONAS
En 1884, Albrecht Kossel reportó el aislamiento de un componente extraído por tratamiento ácido de núcleos de eritrocitos de ganso. Por su aparente similitud fisicoquímica con la peptona lo denominó histonas y sugirió que podría estar unido a los ácidos nucleicos . La palabra histona deriva de la palabra alemana “Histon”, de origen incierto pero probablemente del griego “histanai” o de “histos”. Sólo después de la década de 1990 es que fue reconocido el papel regulador de las histonas, antes eran solamente vistas como matriz para el enrolamiento del material genético (ADN).
Las histonas son proteínas globulares, de baja masa molecular, muy conservadas evolutivamente entre los eucariotas y en algunos procariotas. Forman la cromatina junto con el ADN, sobre la base de unas unidades conocidas como nucleosomas. La cromatina resuelve el problema de restricción de crecimiento de ADN y nucleo, la cromatina esta formada por DNA y proteinas, la principal proteína formadora son las HISTONAS
Las proteínas celulares más frecuentes son las proteínas histonas, siendo que cada célula eucariótica presenta varios cientos de millones de moléculas de histonas, mientras que las demás proteínas no alcanzan unos cientos (como mucho, a miles). Son proteínas de masa molecular baja, aproximadamente 11-12 Kd y exhiben un alto contenido, cerca de 20%, de lisina y arginina (aminoácidos básicos). Con las cargas positivas de las cadenas laterales de estos restos, las histonas (que son extremadamente básicas) se unen a los grupos fosfato del ADN (cargados negativamente); para ello no es relevante la secuencia de bases dentro del ADN. A menudo, las histonas son modificadas por metilaciones, acetilaciones, fosforilaciones o ADP-ribosilaciones.
–Metilaciones: Determinan cambios permanentes en la cromatina. Están destinadas al mantenimiento de un tipo determinado de expresión génica (Enzimas encargadas: HMTasas)
–Acetilaciones: en las colas de las histonas a nivel de lisina y arginina: modifican la cromatina determinando que pueda transcribir (Enzimas encargadas: HAT)
–Desacetilaciones: determinan la compactación de la cromatina, silencia la actividad transcripcional (Enzimas encargadas: HDAC)
En los seres humanos hay cinco tipos principales: la histona H1 y las histonas H2A, H2B, H3 y H4. Estas últimas se denominan también histonas nucleosomales y forman un octámero con dos histonas de cada; alrededor de este núcleo se enrolla dos veces un hilo de ADN. Este complejo ADN-histona recibe el nombre de nucleosoma y constituye el componente primario del cromosoma.
–Metilaciones: Determinan cambios permanentes en la cromatina. Están destinadas al mantenimiento de un tipo determinado de expresión génica (Enzimas encargadas: HMTasas)
–Acetilaciones: en las colas de las histonas a nivel de lisina y arginina: modifican la cromatina determinando que pueda transcribir (Enzimas encargadas: HAT)
–Desacetilaciones: determinan la compactación de la cromatina, silencia la actividad transcripcional (Enzimas encargadas: HDAC)
En los seres humanos hay cinco tipos principales: la histona H1 y las histonas H2A, H2B, H3 y H4. Estas últimas se denominan también histonas nucleosomales y forman un octámero con dos histonas de cada; alrededor de este núcleo se enrolla dos veces un hilo de ADN. Este complejo ADN-histona recibe el nombre de nucleosoma y constituye el componente primario del cromosoma.
El ADN gira unos 147 pares de bases alrededor del núcleo de la histona y a continuación se desplaza unos 20-70 bp en un giro hacia la izquierda hasta alcanzar el siguiente nucleosoma. La pieza intermedia, también denominada ADN de conexión está “desnuda”, es decir, no está equipada con histonas. La histonas H1 se coloca como pieza de cierre en cada nucleosoma y al mismo tiempo toma contacto con las agrupaciones vecinas. De esto modo, las proteínas H1 van “grapando” los nucleosomas para formar un hilo denso: la fibra de cromatina.
Suscribirse a:
Entradas (Atom)




























